据大通福克斯美国会诊中心(CMAA)了解:本周《细胞》封面聚焦了泛癌症图谱研究在一项长达十余年的研究工作中,癌症研究人员对包含33种主要癌症的1万份癌症病例进行了分析和重新分类,并绘制出一份有史以来最大规模的“泛癌症图谱”。最新相关资讯信息,大通福克斯美国会诊中心(CMAA)将持续为您报道。
这项工作颠覆了人类对癌症分类的传统认识,为我们理解癌症如,何产生、如何治愈癌症提供了重要指导。谈到癌症的种类,我们想到的是胃癌、乳腺癌、肝癌等——的确,一般来说,人们基于癌症在体内的起源部位来对其进行分类。
但是,2012年在美国加州圣克鲁斯的一次会议上,研究人员发起了名为“泛癌症倡议”(the Pan-Cancer Initiative, PCI)的合作项目,试图从一个全新的角度——分子层面来研究癌症。初步研究表明,起源于不同器官的癌症实际上在分子层面可能具有诸多共性,而来自同一组织的癌症却可能有着截然不同的基因组图谱。
如今,基于对涵盖了33种主要癌症类型的1万多份癌症案例的分析,PCI项目公布了更大规模的基因组与分子数据分析结果。这项由美国国家癌症研究所(the National Cancer Institute, NCI)和美国国家人类基因组研究所(the National Human Genome Research Institute, NHGRI)合作取得的分析结果又被称为“泛癌症图谱”(the Pan-Cancer Atlas),是迄今为止最全面的跨癌症分析,也是癌症基因组图谱(The Cancer Genome Atlas, TCGA)项目的最终成果。
研究结果已于4月5日通过27篇论文发表在《细胞》(Cell)、《癌细胞》(Cancer Cell)、《细胞报道》(Cell Reports)和《免疫》(Immunity)期刊上。
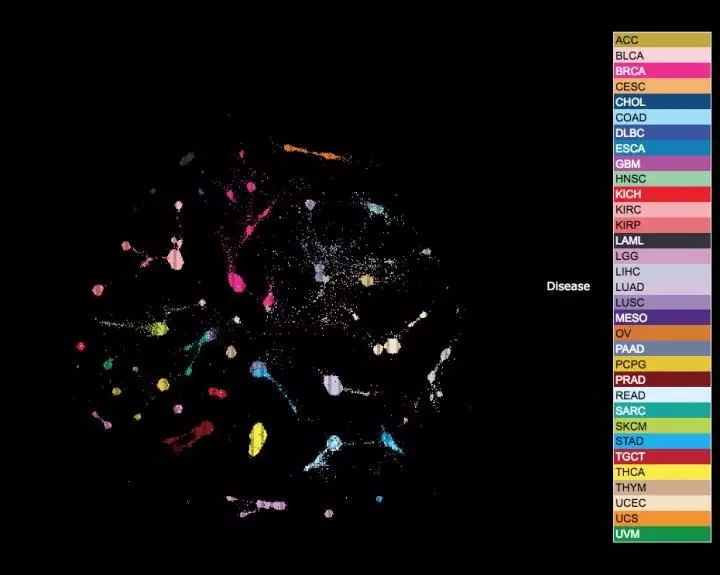
UCSC肿瘤地图(the UCSC Tumor Map)帮助研究人员将从TCGA数据中发现的主要模式可视化,包括细胞来源、分子组织学、“干性”或分化状态、特异性改变的遗传特性……(来源:加州大学圣克鲁斯基因组研究所)
癌症的新分类
“这项研究对于某种癌症如何与另一种疾病相关联的见解具有实际临床意义”,加州大学圣克鲁斯分校的生物分子工程学教授、PCI组织者乔什·斯图尔特(Josh Stuart)说,“在这种情况下,我们就可以从更熟悉的疾病那里借鉴医疗手段,并将其应用于我们还不甚了解的癌症。”
巴克衰老研究所(Buck Institute for Research on Aging)的癌症与启发性疗法教授、加州大学旧金山分校(UC San Francisco)临床肿瘤医师克里斯托弗·本茨(Christopher Benz)和巴克研究所高级研究员、加州大学旧金山分校外科助理教授克里斯蒂娜·邱(Christina Yau)是斯图尔特泛癌症分析项目中的主要合作者。斯图尔特和本茨在加州大学布鲁克斯分校-巴克研究所基因组数据分析中心(UCSC-Buck Institute Genome Data Analysis Center)担任联合主任,在TCGA项目的研究机构网络中,全美共有7处这样的国家级数据分析中心。
研究人员开展国际合作,对完整的TCGA肿瘤数据集进行了全面的分子层面分析。分析结果表明,无关乎每种肿瘤的解剖学位置,可根据细胞和遗传组成将全部33类肿瘤亚型重新划分为28种不同的分子亚型,或称为“簇”(cluster)。在这些簇中,有近三分之二的肿瘤属于异质性的,因为它们包含了多达25种不同的组织学肿瘤类型。传统意义上,针对这些不同组织学类型的肿瘤需要不同的治疗方案。这些与多种预后模型相关联的分子分析和聚类结果,已通过TCGA门户网站向全世界的临床医师和研究人员开放。
“这一全面的TCGA泛癌症图谱分析将为未来的癌症研究工作和临床试验提供新的理论依据”,本茨说道,“它还将激励临床肿瘤学家使用基因组特征来划分新诊断出的和复发的肿瘤。如果可以根据基因组和分子层面的组成来对患者的肿瘤进行鉴别和分类,就能帮助他们找到最佳治疗时机。”
跨肿瘤比对的第一轮研究于2013年9月完成,当时斯图尔特及其在PCI项目的同事分析了TCGA项目提供的12种肿瘤图谱。尽管大多数肿瘤最终都可以根据其组织来源进行分组,但斯图尔特等人发现,在不同组的肿瘤中仍有许多共同的DNA、RNA和蛋白质信号。
“(那时)一切都变得明朗起来了,当我们发现不同类型的癌症仍有相似之处时,自然就会想做一个更全面的比较。”斯图尔特说。
2014年,斯图尔特合著了另一篇泛癌症研究论文,在那篇论文中,研究人员使用统计学方法对肿瘤分子数据进行分析,将肿瘤重新划分为不同的亚型(或称为簇)。尽管大多数癌症亚型都与其组织起源相匹配,其中仍有一些簇是由起源于身体不同部位的肿瘤组成的。2014年的研究表明,如果采用新的分子分类方法,每10名癌症患者中就有1人会被归为与原先认知不同的癌症类型,而这些差异可能会对该为患者提供何种治疗方案或临床试验产生影响。
将研究推向临床
作为TCGA项目和国际癌症基因组学联盟(International Cancer Genomics Consortium)泛癌症工作组联合主任,斯图尔特在确保合作研究在统一规则下有序进行、并由同一个指导委员会监督方面起到了关键的作用。
斯图尔特协助组织了4月5日在细胞出版社(Cell Press)下属期刊上发表的27篇泛癌症图谱论文。他说,“泛癌症”计划迄今为止的成果就像一棵巨大的树,主根代表着不同的肿瘤分类方法,而从主根上又可以分出许多不同的根系。这种组织结构为“主题化类别”划分提供了基础,而非以往基于器官或组织类型的划分。

斯图尔特和本茨是其中一篇《细胞》研究论文的主要作者,该论文的研究工作由美国文安德研究所(Van Andel Research Institute)的彼得·莱尔德(Peter Laird)主导,其成果为其他希望深入研究各类别数据的研究人员提供了一幅路线图。“我们的调查试图找出数据背后隐藏了哪些系统。相比于临床意义,更重要的是我们从中发现的模式”,斯图尔特说。论文的第一作者包括邱和斯图尔特位于加州大学圣克鲁斯基因组研究所的实验室的在职科学家克里斯托弗·王。
斯图尔特说,他十分确信,一旦科学家们开始审视这些数据,它们带来临床价值的日子也就不远了。“很显然,从这些‘根系’中找到可操作的逻辑就像寻找圣杯,”他说,“我们在这篇论文中曾立下里程碑,而如今我们终于能够后退一步,看清这幅完整的蓝图。”
由尤利娅·牛顿(Yulia Newton)和亚当·诺瓦克(Adam Novak)开发的交互式网站“UCSC肿瘤地图”(UCSC Tumor Map)帮助研究人员将数据可视化,在谷歌地图般的用户界面上显示患者样本。在发表于《细胞》的论文中,王使用这一网站对从数据中获得的10个主要模式进行了显示。这些模式包括细胞起源、分子解剖学、“干性(stemness)”或分化状态、特异性改变的基因通路和肿瘤的免疫系统成分。
“看着这些肿瘤地图就像首次从卫星轨道上看着地球”,斯图尔特说,“我们现在看到的是癌症的全貌,它让我充满希望,希望我们有朝一日能够理解它有限的特性,而不是无限的复杂性。”
改写癌症治疗策略
加州大学圣克鲁兹分校准备从最受欢迎的UCSC基因组网站开始,为生物学研究创建更多的专门网站。“我们非常愿意通过这个新的门户网站公开提供这些丰富的数据”,斯图尔特说。
本茨说,最新的TCGA数据可能在免疫疗法中格外有前途,比如,一些已获批准的免疫疗法在针对有限数量的传统类型癌症治疗中产生了近乎不可思议的结果。值得注意的是,该研究显示,在观察到的28个不同分子类型簇中,有一个簇由25种不同的传统类型肿瘤组成,并表现出非常强的与激活患者免疫反应相关的特征。
“这一发现再次支持了一种逐渐占据主导地位的观点,即FDA每批准针对一种癌症类型的特异性免疫疗法,都可能会使其他类型的癌症患者同样受益,只要这些其他类型的癌症能够被同样的分子识别。”本茨说。
批准用于其他疾病的药物,也有可能能够有效对抗一些新分类下的癌症亚型。“两种我们新定义的癌症簇也表现出了JAK/Stat分子途径的激活,这一途径通常会在类风湿性关节炎中上调”,邱说,在过去十年中,她完成了TCGA项目中的大部分生物信息学分析工作,“我们将有可能从以往用于治疗良性慢性疾病的药物中找到新的用途,因为现在研究人员将能够根据分子层面的理论基础来探索新的治疗策略。”
即使TCGA项目已经结束了——不会再对数据库进行更新或修改,但在NCI的资助下,这类综合和协同的多平台基因组分析仍将在全国范围内继续进行。斯图尔特、本茨和邱还将继续为新的基因组数据组成分析网络项目开展合作。这也将带来新的挑战:确定临床可测量的生物标志物,以使其更容易和更有效地识别由TCGA网络的多平台分析鉴别出的相同肿瘤的分子集。
“现在是时候改写教科书中对癌症分类的描述了,也该打破临床肿瘤学领域中阻碍患者受益于新癌症分类模式的障碍了”,本茨说。
原文链接
https://www.eurekalert.org/pub_releases/2018-04/uoc--na040418.php
大通福克斯美国会诊中心(CMAA)是一家专注于为中国患者提供美国高端精准医疗咨询和服务的专业机构。自成立至今,已经为许许多多的中国患者提供了这样的服务,不仅让他们体会到了高端的,专业的服务,而且接受了精准的,个体化的治疗。最重要的是,通过中心提供的服务,很多病人获得了满意的,甚至是超越他们期望的治疗效果。
▌ 推荐阅读
1、电脑链接 ▪ 手机连接
BET抑制剂将问世?新分子有望双重防治癌症
2、电脑链接 ▪ 手机连接
FDA今日加速批准首款特定白血病疗法,有望彻底消除病根
3、电脑链接 ▪ 手机连接
FDA今日批准新款HIV疗法,可低成本治疗感染






